“Rosetta@home”的版本间差异
小 |
小 (更新开发者国旗) |
||
| 第5行: | 第5行: | ||
| caption =Rosetta@home 的屏幕保护图形 | | caption =Rosetta@home 的屏幕保护图形 | ||
| developer =华盛顿大学贝克实验室 | | developer =华盛顿大学贝克实验室 | ||
| − | Rosetta Commons | + | Rosetta Commons[[Image:United_States.gif]] |
| released =2005年6月26日 | | released =2005年6月26日 | ||
| − | | operating system =[[Image:Windows.png]][[Image:64bits.png]]/[[Image:Linux.png]][[Image:64bits.png]]/[[Image:Macos.png]] | + | | operating system =[[Image:Windows.png]][[Image:64bits.png]] / [[Image:Linux.png]][[Image:64bits.png]] / [[Image:Macos.png]] |
| platform =[[BOINC]] | | platform =[[BOINC]] | ||
| program info =Rosetta Mini≈345.4MB | | program info =Rosetta Mini≈345.4MB | ||
2010年9月3日 (五) 17:41的版本
|
Rosetta@home | |
|---|---|
| 230px Rosetta@home logo | |
 Rosetta@home 的屏幕保护图形 | |
| 开发者 | 华盛顿大学贝克实验室 |
| 版本历史 | 2005年6月26日 |
| 运算平台 | |
| 项目平台 | BOINC |
| 程序情况 | Rosetta Mini≈345.4MB |
| 任务情况 | 需要400MB左右硬盘空间、任务可以自行设定1至24小时的运算时间、任务限期10天 |
| 项目状态 | 运行中/开放注册 |
| 项目类别 | 生命科学类 |
| 优化程序 | 无 / 64位计算程序比32位有效率提升 |
| 计算特点 | CPU密集: |
| 官方网址 | Rosetta@home |
| [{{{rss}}} 通过 RSS 获取项目新闻] | |
Rosetta@home 是一个基于伯克利开放式网络计算平台 BOINC 的分布式计算项目。该项目由华盛顿大学贝克实验室开发和维护,用于蛋白质结构预测、蛋白质-蛋白质对接和蛋白质设计的研究。截至 2009 年 2 月 8 日,全球共有 8.6 万台计算机是这一项目的活跃志愿者,平均执行速度达 78 万亿 FLOPS。Rosetta@Home 还开发了一款电子游戏 Foldit,目的是通过众包(crowdsourcing)途径来实现上述研究目标。尽管这个项目很大程度上侧重于进行提高蛋白质组学方法的精确性和稳固性的基础研究,它也进行一些关于艾滋病、疟疾、癌症、阿兹海默病以及其他疾病的病理学的应用研究。
与其他 BOINC 项目一样,Rosetta@home 使用志愿者的计算机中空闲的进程资源来执行单独的单元计算。计算结果会被发送到项目的中央服务器,经验证后存入数据库中。这个项目是跨平台的,支持多种不同的软件和硬件环境。用户可通过 Rosetta@home 的屏幕保护程序观看正在自己计算机上进行的蛋白质结构预测的情况。
除了疾病相关研究,Rosetta@home 网络还是结构生物信息学中新方法的一个测试框架。这些新方法经 Rosetta@home 庞大且多样的用户群体使用后,若运行效果稳定,将会被用于其他基于 Rosetta 的应用程序,例如 RosettaDock 和人类蛋白质组折叠项目。新方法测试中的两个重要项目是蛋白质结构预测技术的关键测试(CASP)和交互作用预测的关键测试(CAPRI)。这两项测试实验分别用于评估蛋白质结构预测和蛋白质-蛋白质对接预测的最前沿技术。Rosetta@home 稳居最重要的对接预测器之一,并且是现有最好的蛋白质三级结构预测器之一。
如何加入项目
该项目基于 BOINC 平台,简要的加入步骤如下(已完成的步骤可直接跳过):
- 下载并安装 BOINC 的客户端软件(官方下载页面或程序下载)
- 点击客户端简易视图下的“Add Project”按钮,或高级视图下菜单中的“工具->加入项目”,将显示向导对话框
- 点击下一步后在项目列表中找到并单击选中 Rosetta@home 项目(如未显示该项目,则在编辑框中输入项目网址:http://boinc.bakerlab.org/rosetta/ ),然后点击下一步
- 输入您可用的电子邮件地址,并设置您在该项目的登录密码(并非您的电子邮件密码)
- 再次点击下一步,如项目服务器工作正常(并且有适合自身操作系统的计算程序),即已成功加入项目
更详细的加入方法说明,请访问 BOINC 新手指南 或 BOINC 使用教程。
本站推荐您加入 Team China 团队,请访问项目官方网站的 团队检索页面,搜索(Search)并进入 Team China 的团队页面,点击页面中的 Join 并输入用户登录信息即可加入!
计算平台
Rosetta@home 应用程序和 BOINC 均支持 Microsoft Windows、Linux 和苹果机平台。(BOINC 还可在更多平台上运行,如 FreeBSD。)参与 Rosetta@home 的客户端计算机需要有一个时脉速度至少达到 500 兆赫的中央处理器、400MB 空余的硬盘空间、512MB 的物理内存,以及因特网连接。截至2009年5月29日,Rosetta 应用程序的最新版本号是5.98,Rosetta Mini 应用程序的最新版本号是 1.71。用户的 BOINC 客户端与位于华盛顿大学的 Rosetta@home 服务器端之间使用标准 HTTP(80端口)进行通信,HTTPS(443端口)用于密码交换。BOINC 客户端使用 1043 和 31416 端口进行远程和本地控制,这两个端口可能需要在防火墙中被设置为“解除封禁”才可被使用。包含蛋白质数据的工作单元由服务器分配给志愿者的计算机(客户端),然后客户端对所分配得到的任务进行蛋白质预测运算。为了避免重复的预测,每个工作单元会得到一个初始的随机种子。这使得每个预测具有独一无二的沿蛋白质能量图景(energy landscape)的下降轨道。对于给定的蛋白质能量图景,Rosetta@home 的结构预测近似为整体极小值。这个整体极小值代表该蛋白质的能量最佳构造,即它的自然态。
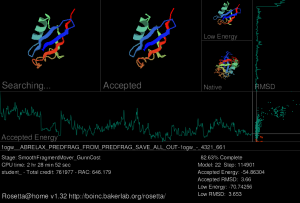
Rosetta@home 的图形用户界面是一个屏幕保护程序,显示了当前工作单元进行蛋白质折叠模拟的情况。屏幕左上方为当前蛋白质链正在尝试的移动(即搜索的形状)。紧邻其右侧是最新一个被接受的移动。再往右侧分上下两个小图,上图为当前最低能量形状,下图为实验中得到的真实形状(如果已知)。屏幕中部显示的是被接受模型的自由能变化曲线。屏幕上方最右侧是接受模型的均方根偏差(RMSD)曲线,体现了被接受模型与实验中真实模型之间的相似度。在自由能变化曲线右侧、RMSD 曲线下方,使用这两项结果生成了一个能量 /RMSD 图,伴随着模型的不断精确。
与所有其他 BOINC 项目类似,Rosetta@home 利用客户端空闲资源,在后台执行。执行过程可能发生在用户登录操作系统之前或之后。当其他应用程序需要时,Rosetta@home 会释放资源,因此不会影响用户对计算机的正常使用。为了使计算机的功耗或者放热降到最低,用户可以自行指定 Rosetta@home 使用 CPU 资源的最大比例。此外,Rosetta@home 每天最多运行次数,以及更多选项也都可以通过用户的账户选项来进行设置。
Rosetta@home 网络所使用的 Rosetta 软件最早用 Fortran 编写,后改用 C++ 重新编写,以利于进一步的开发。新版本于2008 年 2 月 8 日发布,实现了面向对象。Rosetta 代码由 Rosetta Commons 开发。这个软件对学术使用免费,对制药公司则收取费用。
项目意义
通过一系列的基因组测序计划,科学家能够判定许多种在细胞内发挥作用的蛋白质的氨基酸序列或者一级结构。为了更好地了解蛋白质的功能,以及向合理化药物设计提供辅助,科学家们需要知道蛋白质的三级结构。
蛋白质的三维结构目前主要通过X射线晶体学或核磁共振技术来进行实验确定。这个过程十分耗时,例如可能花费数星期或者几个月才能首次研究出如何使一种蛋白质结晶;而成本又非常高,每种蛋白质耗费约 10 万美元。更重要的是,发现新序列的速度远超确定结构的速度:美国国家生物技术信息中心非冗余蛋白质数据库中存在的超过 740 万个蛋白质序列中,仅有不到 5.2 万已被确定结构并被存入蛋白质数据库中。Rosetta@home 的一个主要目标是在显著降低时间和金钱成本的情况下,预测蛋白质结构,并且达到与现有实验方法同样的精度。Rosetta@home 还开发了确定膜蛋白(如G蛋白偶联受体)结构和对接的方法。膜蛋白是现代药物设计的主要目标,但通过X射线晶体学、核磁共振等传统技术却极难获得其结构。
蛋白质结构预测的进展通过两年一届的蛋白质结构预测技术的关键测试(CASP)实验来进行评估。在这项实验中,来自全球各地的研究人员尝试从氨基酸序列中得到蛋白质结构。这项实验有时竞争十分激烈,得分高的小组被认为是最高端蛋白质结构预测研究的事实上的标准制定者。Rosetta@home 所基于的 Rosetta 程序,自 1998 年的 CASP3 实验上开始被使用。在 2004年的 CASP6 上,Rosetta 创造了历史,在它为 CASP 目标蛋白质 T0281 提供的模型中,首次生成了接近原子级精度的 ab initio 蛋白质结构预测。Ab initio 不使用结构同源的信息,而必须依赖于序列同源信息以及蛋白质内的模拟物理交互,因此被认为是一类特别难以预测的蛋白质结构。Rosetta@home 自2006年 CASP7 上开始被使用。在 CASP7 上,它在每个类别的预测中都成为最好的预测器之一。而高质量的预测需要来自 Rosetta@home 众多志愿者提供计算资源。不断增多的计算资源使Rosetta@home 能够对构象空间(一个蛋白质可以被假设具有的可能的形状)更多的区域进行取样,根据“黎文索尔佯谬”(Levinthal paradox),采样数量会随着蛋白质长度的增长而呈指数增长。
Rosetta@home 也被用于蛋白质-蛋白质对接预测。这项预测确定蛋白质复合体结构或者四级结构。这一类型的蛋白质交互作用影响到许多细胞功能,包括抗原-抗体、酶-抑制剂捆绑等。确定这些交互作用在药物设计中十分关键。Rosetta 被用于交互作用预测的关键测试(CAPRI)实验。这项实验评估当前最前沿的蛋白质对接技术,评估模式与 CASP 类似。Rosetta 在这项实验中得到的结果属最精确、最完整之一,而志愿者提供的计算资源被认为是 Rosetta 获得成功的主要因素之一。
2008年初,Rosetta 被用来推算设计一种具有在自然界中从未被观察到功能的蛋白质。这个灵感源自2004年一篇引起关注的、被撤稿的论文,那篇论文中描述了一种蛋白质的推算设计,这种蛋白质与天然的蛋白质相比提高了酶活力。2008年,大卫·贝克的研究组发表了论文,描述了这种蛋白质的制造过程。论文指出 Rosetta@home 为其提供计算资源,作为这种蛋白质设计方法的一项重要的概念论证。这一类型的蛋白质设计将来可能在药物设计、绿色化学、生物修复等领域得到应用。
与类似的分布式计算项目的比较
目前有多个分布式计算项目与 Rosetta@home 具有类似的研究方向,但研究方法上存在差异。
Folding@home
斯坦福大学开发的 Folding@home 是与蛋白质研究相关的主要分布式计算项目中唯一不使用 BOINC 平台的。Rosetta@home 与 Folding@home 都研究蛋白质错误折叠疾病(如阿兹海默病),但 Rosetta@home 还进行其他研究,而 Folding@home 则主要集中于这类研究。Folding@home 并不采用基于结构或者基于设计的方法来预测淀粉体行为,而是采用分子动力学方法来构建蛋白质折叠活动(以及可能的错误折叠和聚合)的模型。换言之,Folding@home 的优势在于模拟蛋白质折叠活动,而 Rosetta@home 的优势则在于蛋白质运算设计以及蛋白质结构和对接的预测。这两个项目在计算资源和主机分布上也存在显著差异。Rosetta@home 的主机群体基于 PC,平均执行速度为 78 万亿 FLOPS;而 Folding@home 的主机群体包括了 PlayStation 3 和图形处理器,平均执行速度达 4769 万亿 FLOPS,大约是 Rosetta@home 的 61 倍。
World Community Grid
World Community Grid(世界公共网格)的子项目人类蛋白质组折叠项目(HPF)1 期和 2 期均使用 Rosetta 程序来为不同的基因组添加结构和功能注解。人类蛋白质组折叠项目的首席科学家里夏尔·博诺在华盛顿大学贝克实验室攻读博士学位期间积极参与了 Rosetta 的早期开发,但他现在主要使用 Rosetta 来为生物学家创建数据库。他的个人网站上设置了关于 HPF1、HPF2 的信息布告板。
Predictor@home
与 Rosetta@home 相似,蛋白质结构预测也是 Predictor@home 的研究重点。Predictor@home 还计划在其分布式计算平台上开发蛋白质设计与对接的新研究领域(采用分子动力学的 CHARMM 软件包)。这将使它与 Rosetta@home 更加相似。进行结构预测时,Rosetta@home 使用的是 Rosetta 程序,而 Predictor@home 则使用 dTASSER 方法。
其他
BOINC 平台上其他的蛋白质相关分布式计算项目包括 QMC@Home、Docking@home、POEM@home、SIMAP 和 TANPAKU。RALPH@home 是 Rosetta@home 的 alpha 版本,用来进行新应用程序、工作单元以及更新被添加到 Rosetta@home 之前的测试工作。RALPH@home 也在 BOINC 平台上运行。
志愿者的贡献
Rosetta@home 的研究依赖于大量志愿者提供的计算资源。截至 2009 年 1 月 9 日,Rosetta@home 的活跃用户超过 4.5 万人,分布在 168 个国家,总共提供了 8.2 万台计算机的空闲资源,使 Rosetta@home 的平均执行速度达到 73 万亿FLOPS。[3]
用户的贡献通过 BOINC 积分来度量。一个用户从某一工作单元得到的积分是他在这个工作单元上产生的“诱饵”数量与所有用户在该工作单元获得的积分均值的乘积。Rosetta@home 对 CPU 每秒工作所给的积分低于绝大多数其他 BOINC项目。尽管如此,Rosetta@home 仍在所有 BOINC 项目中名列总积分值第 5 位。
预测的蛋白质结构被提交到 CASP 实验的用户,也在相关的学术出版物中被致谢。而预测出指定工作单元最低能量结构的用户及其所在队伍则会在 Rosetta@home 的主页上被列为“当日预测者”(Predictor of the Day)。每天还有一名随机选出的用户会被列在主页上,成为“当日用户”(User of the Day)。
继续阅读
- Rosetta@home:David Baker 的欢迎信
- Rosetta@home:常见科学问答
- Rosetta@home:相关疾病的研究
- Rosetta@home:发展历史与分支
- Rosetta@home:屏幕保护
相关链接
- 中国分布式计算论坛 Rosetta@home子版块
- Rosetta@home项目官方网站
- 大卫·贝克的Rosetta@home日志
- BOINC网站上关于Rosetta@home的统计数据
- RALPH@home项目网站
- Rosetta Commons网站
- Robetta项目
- RosettaDesign项目
- RosettaDock项目