Folding@home
Folding@home 是一个研究蛋白质折叠、误折、聚合及由此引起的相关疾病的分布式计算工程。由斯坦福大学化学系的潘德小组(Pande Group)主持,于2000年10月1日正式启动。Folding@home 目前是世界上最大的分布式计算项目,于2007年为吉尼斯世界记录所承认。
2004年3月8日,研究基因结构的 Genome@home 计划终止,并入 Folding@home。

项目意义和研究成果
Folding@home 注于精确地模拟蛋白质折叠和错误折叠的过程,以便能更好地了解多种疾病的起因和发展,包括阿兹海默症(脑退化症)、牛海绵状脑病(疯牛症)、多种癌症和癌症相关综合症、囊胞性纤维症。到目前为止,Folding@home 可成功模拟长达5秒时段的折叠过程,超出先前估计可模拟的时段数百万倍。
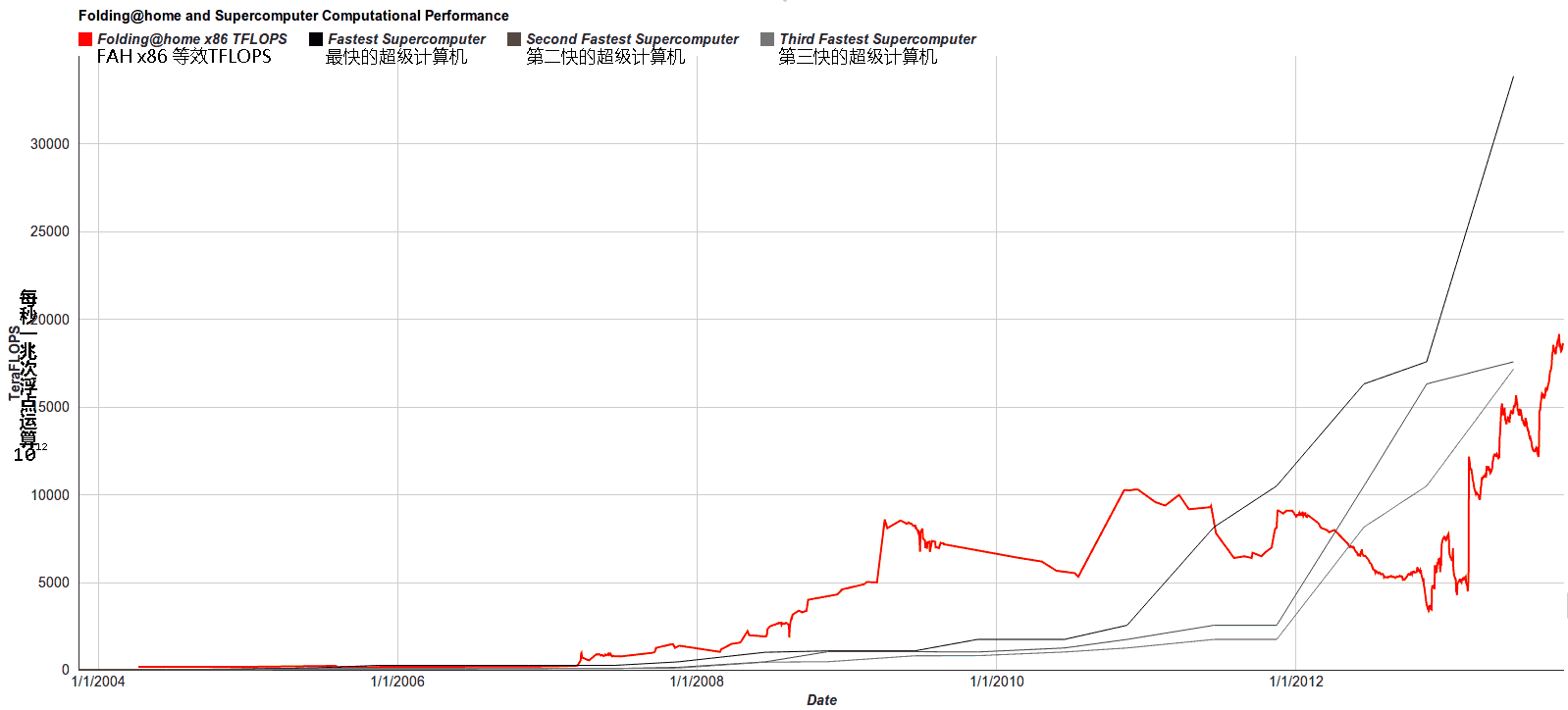
正如大家所知,Folding@home项目目前是世界上最大的分布式计算项目,于2007年为吉尼斯世界记录所承认,截止目前有超过百万人参与项目并提交成果,它的计算能力总和也能达到全球超级计算机TOP10水平。同时该项目也是AMD和NVIDIA等GPU厂商最早参与推进的分布式计算项目,这让项目进程大大加快,用户参与更加方便。如图所示:Folding@home项目汇聚的计算能力,已经超越2012年底,全球排名第二的超级计算机。
什么是蛋白质?它们是怎么折叠的呢?
蛋白质是一个生物体系的网络基础,它们是一个个纳米级计算机。在蛋白质实现它的生物功能之前,它们会把自己装配起来,或者说是折叠;虽然蛋白质折叠对所有的生物来说是最基本的和最明确的事实,但它的折叠过程对人类而言仍然是个未解之谜。
此外,当蛋白质没有正确的折叠(误折)无疑会产生严重的后果,包括许多知名的疾病,比方阿兹海默症(Alzheimer's disease/脑退化症),疯牛病(Mad Cow, BSE),可传播性海绵状脑病(CJD),肌萎缩性脊髓侧索硬化症(ALS),还有帕金森氏症(Parkinson's),多种癌症和其相关综合症(many Cancers and cancer-related syndromes)。
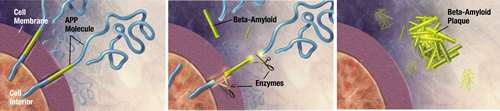
例如:阿兹海默症(Alzheimer's)
比如阿尔茨海默病,这是一种无法治愈的神经退行性疾病,通常影响老年人占一半以上的痴呆症。其确切原因尚不清楚,但疾病被确定为蛋白质的错误折叠疾病。这种病的致命机理是:β淀粉样蛋白(一种有毒的聚合相关的β)肽,由β错误折叠和聚集在一起和其他β肽而引起。图片是与β-淀粉样蛋白片段在脑中的聚集(右)。研究人员利用Folding@Home来模拟这种聚集的过程中,为了更好地理解这种疾病的原因。
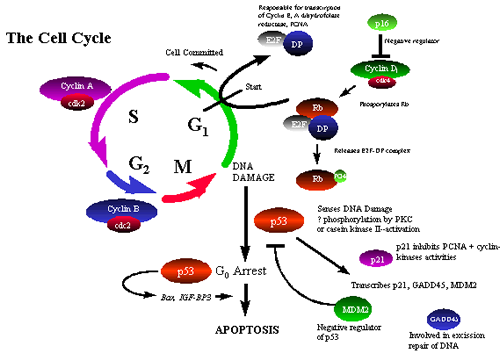
例如:癌症(Cancers)
一半以上的所有已知的癌症都涉及到突变的p53基因,肿瘤抑制蛋白中的每一个细胞,调节细胞周期和信号在DNA损伤的细胞死亡的事件的现。在p53突变可以破坏这些功能,允许一个异常细胞继续生长抑制,导致肿瘤的发展。这些突变分析有助于解释p53相关癌症的根本原因。
相关文章,请参考:Folding@Home项目科学意义与原理简述

Folding@home项目硬件性能体现与影响
Folding@home的客户端利用了经修改的TINKER、GROMACS、AMBER及CPMD这四款分子模拟程式进行运算,并会在许可的情况下作出优化,以把运算速度加快。2006年9月底,ATI宣布了通用计算GPGPU架构,并得到了斯坦福大学Folding@Home项目的大力支持,加入了人类健康研究。2007年3月22日,PS3正式加入史丹佛大学分布式运算研究计划,至今已有超过百万名 PS3 玩家注册参与。NVIDIA于2008年6月宣布旗下基于G80及以上核心的显卡产品都支持该项目的通用计算,更是对分布式计算的重要贡献。
Folding@home项目由于其高密度分子动力学计算原理,对于CPU、GPU等硬件的资源消耗是非常明显的,因此它也几乎成为芯片性能测试的应用类项目之一。基于GPU的计算从概念上讲很容易理解,并且现有多种高级语言和软件工具可以简化GPU的编程工作,不过对于普通用户来说,通过该项目体验GPU计算是最合适不过的。
由于计算分子之间长程力的影响,Folding@home项目计算代码中代码条件分支也随处可见,对GPU Shader灵活度的要求很高,Folding@home在GPU使用量上也要大于图形编程。客观上讲,NVIDIA凭借GeForce 8800GTX业界首个DirectX 10 GPU,在GPU通用计算方面实现了大步跨越,而AMD的ATI Radeon X1900却是并行计算在民用级别第一次大规模推动的代表作。
Folding@home项目对于GPU的考验,主要集中在流处理器的计算自由度方面,这就迫使GPU具备更强大的调度能力、缓存体系。
NVIDIA方面,G80架构下的8000/9000系列产品拥有CUDA并行计算思想和架构的支持,从硬件方面改变了GPU在计算领域的地位。GT200相对于G80也只是强化了并行计算架构,虽然对流处理器的排列组合做了一些微调,但整体架构没有本质变化。到了DirectX 11时代,Fermi架构和之后的架构在Cache、SM架构等等方面做了改革,目的是让GPU核心适应现在甚至未来的用户应用需求。
AMD方面,自从R600架构Radeon HD2000系列以来,AMD的图形构架就没有发生过本质的变化。但是Radeon HD7970的Tahiti构架中,AMD做出了多种巨大的革新和改进,不仅抛却了VLIW带来的种种困扰,几乎完全解决了单元复用率低落的问题,GPU并行计算性能得到释放。
如果你没有条件或者没有必要购买超级计算机,又非常希望体验GPU运算带来的快感,参与体验分子动力学原理的生命科学类Folding@home项目就是最好的选择。
你可以做什么?
你可以下载并运行我们的客户端程序。下载安装运行Folding@home客户端,让你的CPU和GPU不再停歇,让人类距离重大疾病的攻克进程不断推进。加入本项目的计算机越多,本项目计算的速度就越快。
Folding@home 是一个分布式计算的项目—从世界各地来的人下载运行我们的客户端程序,彼此组合在一起构成了世界上最大的超级计算机之一。 每一台参与的计算机都使我们的项目更接近成功一步。Folding@home把分布式运算和革新的计算方法有机的结合在一起,使我们能解决比以前碰到的困难无数倍的问题。
期待您能成为我们的一员:中国志愿计算者名单 和 爱心捐赠者名单
Folding@home是如何工作的呢?
Folding@home是一个研究研究蛋白质折叠,误折,聚合及由此引起的相关疾病的分布式计算工程。我们使用联网式的计算方式和大量的分布式计算能力来模拟蛋白质折叠的过程,并指引我们近期对由折叠引起的疾病的一系列研究。
Folding@home项目属于一般的蛋白质折叠模拟,它仅涉及分子动力学,通常都是把原子视为刚体,然后计算这些刚体之间的力学关系。Folding@home项目的热力学参数通常只有温度,会影响反应进程,但这些过程在folding里通常会被直接转化成力学关系的变化。归根结底,Folding@home项目基于GROMACS计算引擎的,是一个力学主导的分子动力学模拟,它的特点就在于支持大规模分布式计算。
Folding@home所研究的是人类最基本的特定致病过程中蛋白质分子的折叠运动。项目的核心原理,在于求解任务目标分子中每一个原子在边界条件限制下由肽键和长程力等作用所导致的运动方程,进而达到实现模拟任务目标分子折叠运动的目的。每一个原子背后都附庸这若干个方程,每一个方程都可以转换成一组简单的向量指令。
参与该项目的注意事项
- 本项目可以在多平台下进行计算,包括PC、MAC,硬件方面可以支持CPU和GPU加速。
- 最新版的V7综合客户端同时支持多核CPU和AMD/ATI、NVIDIA 系列显卡加速。
- 为获得更好的计算效能,请使用V7综合客户端
- 新手请参考 CPU/GPU参与《新手指南》
- 本项目中国团队的编号为 [3213],欢迎加入中国团队
本项目V7客户端需要硬件、软件列表
- CPU运行V7客户端:
需要的操作系统——Windows XP SP3以上, 32位或64位。推荐使用Win7操作系统。 需要的CPU——Intel P4 1.4 GHz以上,AMD全系列X86架构CPU。推荐使用主频在2GHz以上的多核心处理器,性能更佳。 需要的软件——Microsoft .NET 2.0或更高(WinXP用户)。推荐使用最新。FAH控制界面需要安装2008 C++ Redistributable,否则无法运行 (32位WinXP系统用户) 。 驱动程序——所有硬件驱动程序(特别是显卡)已经安装,并保持较新的版本
与类似的分布式计算项目的比较
目前有多个分布式计算项目与Folding@home具有类似的研究方向,但研究方法上存在差异。
参与Floding@home项目
- Folding@home官方网站首页
- Folding@home项目 CPU/GPU参与《新手指南》
- V7/综合客户端官方下载页面(同时支持CPU&GPU)
- Folding@home(F@H) 简介、各类教程、绿色程序等资料大全
- 参与本站Folding@home项目讨论