“POEM@HOME”的版本间差异
(项目介绍翻译) |
(→'''Project Detail''': 翻译) |
||
| 第25行: | 第25行: | ||
}} | }} | ||
| − | ===''' | + | ==='''项目细节'''=== |
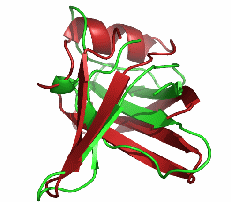
| − | [[Image:Protein-cut.png|right|thumb| | + | [[Image:Protein-cut.png|right|thumb|本图显示了预测的(红色)和实验测得的(绿色)枯草芽孢杆菌主冷激蛋白结构. 这样的蛋白质对实验室条件下的基因调控非常重要.]] |
| − | [http://en.wikipedia.org/wiki/Protein | + | [http://en.wikipedia.org/wiki/Protein 蛋白质] 是所有已知细胞生命的纳米尺度机器. 令人震惊的是, 这些巨大的由多达100,000个原子组成的生物分子还折叠成了独特的三维形状来发挥它们的功能. |
| − | + | 这些功能包括分子化学 ([http://en.wikipedia.org/wiki/Metabolism 新陈代谢]), 能量转换 ([http://en.wikipedia.org/wiki/Photosynthesis#光能到化学能的光合作用]) 和传输 ([http://en.wikipedia.org/wiki/Hemoglobin 氧气运输]), 脑内信号处理 ([http://en.wikipedia.org/wiki/Synapse 神经元]), [http://en.wikipedia.org/wiki/T_cell_receptor 免疫反应] 以及其他众多方面, 而且通常有着任何人工过程无法企及的效率. 蛋白质功能失常往往与疾病相关,目前已经有数千种与疾病有关的蛋白质被鉴定出来,但很多还未确定结构. | |
| − | + | 为了理解,控制甚至是设计蛋白质我们需要研究蛋白质的结构, 而在实验上比获得一个特定蛋白质的化学成分 ([http://en.wikipedia.org/wiki/Protein_sequencing 蛋白质序列])更加困难. | |
| − | + | 通过加入本项目你将为以下研究计算 | |
| − | * | + | *预测蛋白质的生物活性结构 |
| − | * | + | *理解蛋白质相互交流的信号处理机理 |
| − | * | + | *理解与蛋白质功能失常或蛋白质聚集相关的疾病 |
| − | * | + | *在生物学上重要的蛋白质三维结构基础上开发新药物 |
| − | |||
| − | |||
| − | POEM@HOME | + | POEM@HOME 实现了一个理解蛋白质不同方面结构 [http://iwrwww1.fzk.de/biostruct/ProteinFolding/psp_main.htm 重要方法], 并把它提供给[http://boinc.berkeley.edu/ 世界性的分布式计算]. POEM@HOME背后的科学方法是让 [http://en.wikipedia.org/wiki/Christian_B._Anfinsen C. B. 安芬森] 获得1972年诺贝尔化学奖的 [http://en.wikipedia.org/wiki/Anfinsen%27s_dogma 热动力学假说]的计算实现. |
| + | 所以请帮助我们,加入POEM@HOME, 解决上述科学谜团,破解隐藏在蛋白质未知结构中的生物信息. | ||
| + | |||
| + | POEM@HOME是一个致力于提高我们对生物分子结构和功能认识的纯学术,非盈利项目. 所有的结果都会[http://iwrwww1.fzk.de/biostruct/People/ww_pub.htm 发布]在国际同行评审期刊上,并有对POEM@HOME 志愿者的适当注明. | ||
=='''相关链接'''== | =='''相关链接'''== | ||
2010年6月23日 (三) 20:51的版本
|
POEM@HOME | |
|---|---|
POEM@HOME logo | |
无屏保图形 | |
| 开发者 | FZK-INT |
| 版本历史 | 2007年11月13日 |
| 运算平台 | |
| 项目平台 | BOINC |
| 程序情况 | POEM Protein Folding≈76.2MB |
| 任务情况 | |
| 项目状态 | 运行中/开放注册 |
| 项目类别 | 生命科学类 |
| 优化程序 | 无 |
| 计算特点 | CPU密集: |
| 官方网址 | POEM@HOME |
| [{{{rss}}} 通过 RSS 获取项目新闻] | |
POEM@HOME 实现了一个理解蛋白质不同方面结构的重要的方法,并把它提供给世界性的分布式计算 . POEM@HOME背后的科学方法是让 C. B. 安芬森获得1972年诺贝尔化学奖的热动力学假说的计算实现.
POEM@HOME是一个致力于提高我们对生物分子结构和功能认识的纯学术,非盈利项目. 所有的结果都会发布在国际同行评审期刊上,并有对POEM@HOME 志愿者的适当注明.
如何加入项目
该项目基于 BOINC 平台,简要的加入步骤如下(已完成的步骤可直接跳过):
- 下载并安装 BOINC 的客户端软件(官方下载页面或程序下载)
- 点击客户端简易视图下的“Add Project”按钮,或高级视图下菜单中的“工具->加入项目”,将显示向导对话框
- 点击下一步后在项目列表中找到并单击选中 POEM@HOME 项目(如未显示该项目,则在编辑框中输入项目网址:http://boinc.fzk.de/poem/ ),然后点击下一步
- 输入您可用的电子邮件地址,并设置您在该项目的登录密码(并非您的电子邮件密码)
- 再次点击下一步,如项目服务器工作正常(并且有适合自身操作系统的计算程序),即已成功加入项目
更详细的加入方法说明,请访问 BOINC 新手指南 或 BOINC 使用教程。
本站推荐您加入 Team China 团队,请访问项目官方网站的 团队检索页面,搜索(Search)并进入 Team China 的团队页面,点击页面中的 Join 并输入用户登录信息即可加入!
项目细节
蛋白质 是所有已知细胞生命的纳米尺度机器. 令人震惊的是, 这些巨大的由多达100,000个原子组成的生物分子还折叠成了独特的三维形状来发挥它们的功能. 这些功能包括分子化学 (新陈代谢), 能量转换 ([1]) 和传输 (氧气运输), 脑内信号处理 (神经元), 免疫反应 以及其他众多方面, 而且通常有着任何人工过程无法企及的效率. 蛋白质功能失常往往与疾病相关,目前已经有数千种与疾病有关的蛋白质被鉴定出来,但很多还未确定结构. 为了理解,控制甚至是设计蛋白质我们需要研究蛋白质的结构, 而在实验上比获得一个特定蛋白质的化学成分 (蛋白质序列)更加困难.
通过加入本项目你将为以下研究计算
- 预测蛋白质的生物活性结构
- 理解蛋白质相互交流的信号处理机理
- 理解与蛋白质功能失常或蛋白质聚集相关的疾病
- 在生物学上重要的蛋白质三维结构基础上开发新药物
POEM@HOME 实现了一个理解蛋白质不同方面结构 重要方法, 并把它提供给世界性的分布式计算. POEM@HOME背后的科学方法是让 C. B. 安芬森 获得1972年诺贝尔化学奖的 热动力学假说的计算实现.
所以请帮助我们,加入POEM@HOME, 解决上述科学谜团,破解隐藏在蛋白质未知结构中的生物信息.
POEM@HOME是一个致力于提高我们对生物分子结构和功能认识的纯学术,非盈利项目. 所有的结果都会发布在国际同行评审期刊上,并有对POEM@HOME 志愿者的适当注明.
相关链接