Rosetta@home:屏幕保护
Rosetta 及其图像的快速向导
关于 Rosetta
Rosetta 的主要目标之一就是对蛋白质的折叠进行预测。蛋白质是由氨基酸组成的线性复合物分子,常被称之为“链”。氨基酸可以看作是蛋白质“链”上的“节”。这只是一个简单的类比。先来看一条金属链,我们可以任意改变它的形状。如果拉住两边,金属链会变成直线;如果扔在地上,它会随意地变成一个形状。金属链中的每一节都是相同的,而蛋白质却是由二十多种属性(形状,吸引和排斥的作用力等等)各不相同的氨基酸组成的,组合在一起后,各个氨基酸将会相互作用并使得蛋白质链呈现某个特定的形状,我们称之为“折叠”。蛋白质的折叠是由其链中氨基酸的连接顺序所决定的。蛋白质的种类有很多,包含的氨基酸的数量及次序各不相同。
要预测特定蛋白质的形状,我们其实是在寻找具有最低能量的折叠。能量是由很多因素决定的。例如有些氨基酸会由于互相吸引而在空间中靠得近,它们之间的相互作用将导致总体能量的降低。Rosetta 寻找最低能量形状的过程大致如下:
- 从没有任何折叠的链开始(就像一条被拉直的金属链)。
- 移动链中的一部分,产生一个新的形状。
- 计算新形状的能量。
- 根据能量上的变化来决定是否接受这次的移动(否则就放弃)。
- 重复 2~4 的步骤直到链中每一部分都移动了足够多的次数。
我们称上面的过程为一条轨迹,每条轨迹的最终结果就是预测出来的一个结构。Rosetta 会保存每条轨迹中找到的最低能量形状。每条轨迹都是独一无二的,因为每次尝试的移动都是随机决定的。过程中的可能性是如此之多以至于不可能找到同样的最低能量形态。
每条轨迹中都包含两个阶段。第一个阶段中使用简化表示的氨基酸序列以便我们可以快速地尝试各种可能性。本阶段是低分辨率的搜索,在屏保上你会看到蛋白质链大幅度的改变形状。在第二个阶段,Rosetta 使用完全表示的氨基酸序列。这个阶段称之为“弛豫”。蛋白质链不再大幅度移动,为了找到正确的氨基酸排布,它只会尝试小幅度的移动。本阶段是高分辨率的搜索,在屏保上你只会看到蛋白质链的轻微摆动。在一台主流的计算机上,Rosetta 完成第一阶段仅需要几分钟,而第二阶段因为完全表示的氨基酸序列导致的复杂性,将需要花费更长的时间。
对于每一个任务包,你的计算机将要包含 5~20 次的轨迹,然后在完成后将发送给我们每条轨迹中的最低能量形状。我们将检查所有用户计算得到的低能量形状以找到最低的能量形态。这也就是我们对该蛋白质折叠的预测。
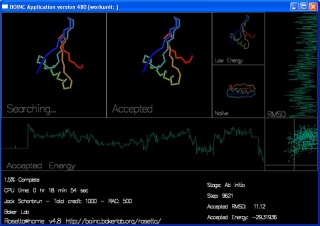
屏保
屏保会显示出每条轨迹正在进行时的进度:
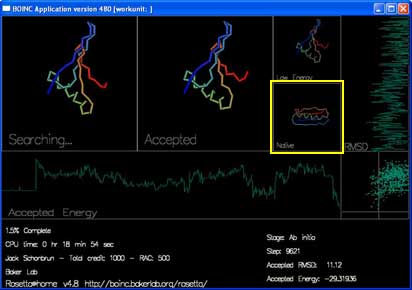
有四个框将显示蛋白质链的形状。
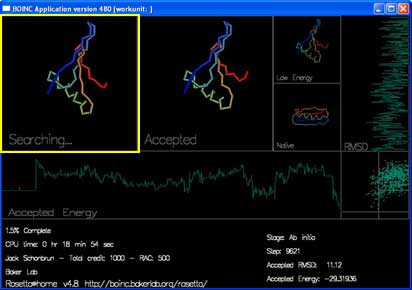
“Searching...” 显示的是当前蛋白质链正在尝试的移动。你可能看到链的形状以及由蓝到红的彩虹颜色。
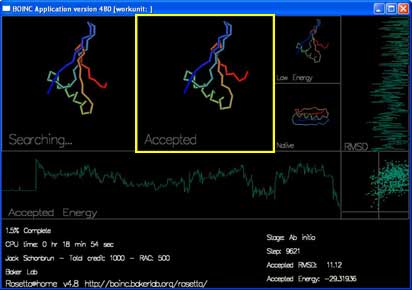
“Accepted” 显示最近被接受的移动。
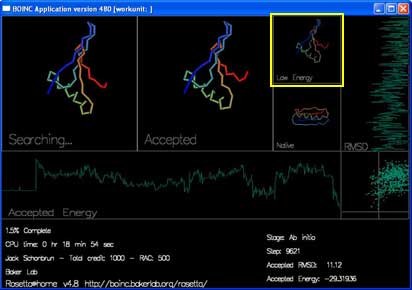
“Low Energy” 显示的是当前轨迹中的最低能量形状。
“Native” 显示的是在实验中得到的真实形状(如果已知的话)。
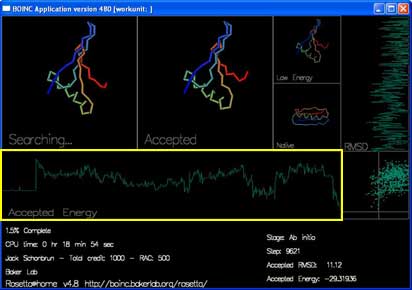
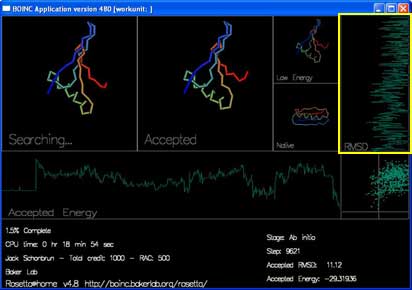
还有两条曲线和一张图即时地显示每次被接受的移动的能量及 RMSD。
“Accepted Energy” 显示的是当前轨迹中每次被接受的移动的能量。(横轴为当前轨迹的进度,纵轴为能量。)
“RMSD” 表示时当前被接受的结构与真实结构的差距。(横轴为 RMSD,纵轴为进度。)
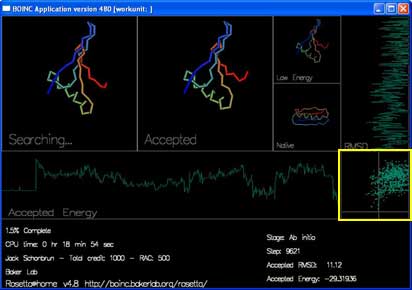
最后一个在屏保右边中间的方框,显示的是每次被接受的移动的能量以及RMSD。这张图和网站上 最佳预测 页面显示的图相类似。不同的是你在屏保里看到的是每条轨迹中每次“被接受”的移动的情况。而在最佳预测页面仅显示每条轨迹的最低能量折叠。