GPUGRID:项目背景
GPUGRID 项目背景
项目介绍
特点
GPUGRID的主要特点是巨大的计算能力,它能获得由CUDA加速的GPU带来的计算能力,不仅仅是计算能力的聚合,更是志愿者个人计算机的聚合。要在一个快速变化的网格中使用宽松的判断准则,那么粒子级是全原子分子模拟的一项基本要求。例如,在一个单一的图形设备上运行分子动力学程序,分析一个由 60000原子构成的分子系统的轨迹,每天只能模拟出4纳秒。在实际应用中,从科学的角度来看,相当于在一个大型个人超级计算机上模拟一个由10万原子构成的分子系统。
目标
我们的目标是执行全原子高通量分子动力学的模拟。尤其是利用大型的计算能力,我们正在建立适应热力学原理的基本分布式计算基础设施,来计算自由能态的蛋白质配体、蛋白质之间的亲和力、筛选取样。这一切无不为更好的理解分子相互影响机制而做的实验提供了更多可能的支持,并能通过一种准确的虚拟筛选过程来促进生物医学研究的发展。
研究内容
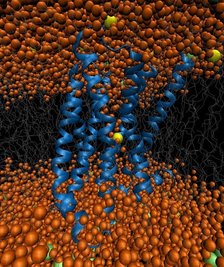
D2多巴胺受体在离子强度作用下的生理反应的分子模拟
钠离子在将D2多巴胺受体融入抗精神病药物合成的过程中扮演着极其重要的角色。了解钠诱导的机制将有助于我们以后设计治疗精神分裂症的药物。 借助分子动力学,我们模拟了流动性钠离子及其动态性能对D2多巴胺受体在离子强度作用下的生理反应。
GPUGRID技术使我们能够操纵一个原子级的微量系统,D2受体被嵌入在一个由61000个原子构成的双层膜中,高性能计算机使得我们能对D2受体进行精确到微秒级的操作。
SH2和多肽配体之间亲合力的分子模拟
SH2是一种由蛋白质之间相互影响形成的蛋白质结构域。这种特殊的结构域在促进细胞生长和发育的信号传导中起着重要的作用。
通过设计更多新的有效的判断规则来确定具有亲和力的蛋白质和多肽,我们将能够在GPUGRID平台上有效地预测成百上千有价值的蛋白质和多肽。
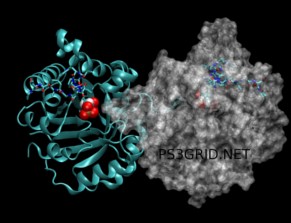
我们正在逐步将需要计算判断亲和力的任务包发放给GPU客户端。
磷酸丙糖异构酶(TPI)的分子模拟
这些任务包用来分析TPI在以下三种情况下的异同:由炎症引发的蛋白质酪氨酸硝化、有缺陷的线粒体呼吸和氧化应激。计算这些任务包通常需要花费一天的时间,并且必须在四天内完成。
前反向指导分子动力学
钾离子弥漫在A型短杆菌肽周围。我们正在发放由30000个原子构成的A型短杆菌肽的全原子模拟的任务包。这些任务包计算花费时间不到一天,并且必须在四天内完成。
计算性能
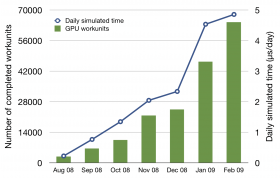
GPUGRID目前正经历一个计算性能飞跃增长的时代。在分子模拟实验中,计算性能可以通过每天的模拟时间总和来衡量。目前,它每天能模拟一个由5万原子构成的分子系统的4~5微秒。
尽管志愿计算能力在不断增加,但是我们依然在不断地努力寻找优化模拟规则的方法,以便我们可以最大收益的运行这个平台。
加速处理器
图形硬件因为处理大量的计算来满足显示逼真的三维图像的需求,其计算能力远远高出标准的处理器。
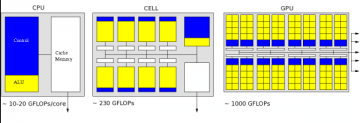
这是通过设计和编程风格的变化来实现的。传统处理器使用相当多的晶体管来实现复杂的逻辑控制,来高性能地执行序列代码。Cell处理器包含8块快速协同处理单元(SPEs),它们可以最大限度的提高算法的吞吐量。图形处理单元(GPUs)设备拥有非常多的并行慢内核,来获得最大化的吞吐量。
例如,仅仅只有一万志愿者就为我们这个星球上某些特定的应用提供了计算能力最大的超级计算机。
关于分子动力学
分子动力学(MD)是一种分子模拟方法,该方法主要依靠近似的已知的物理学,来使用计算机模拟原子和分子体系的运动。由于分子系统通常包含大量的粒子,像这样负责的系统根本不可能找到一套性能分析的方法;分子动力学模拟利用数值计算方法规避了这一问题。它相当于实际试验和理论之间的接口,可以理解为一个“ 虚拟试验”。分子动力学研究分子结构、运动及功能之间的关系。分子动力学是一个多学科的方法。其规则和理论来源于数学、物理和化学,它采用的算法则来自计算机科学和信息理论。分子动力学源于20世界50年代末的物理学,但直到今天,它依然适用于大多数的材料科学和生物分子的模拟。
翻译说明
本页内容翻译自 GPUGRID 的官方网站,由碧城仙翻译。如对本文的翻译有任何建议或疑问,请访问本站论坛的 相关翻译主题。