Rosetta@home:发展历史与分支
Rosetta 最初是作为 ab initio 蛋白质结构预测方法,在1998年由贝克实验室开发;而到目前,这个项目已经形成多个分支,有各自不同的发展和服务方向。Rosetta 平台得名于罗塞塔石碑(被用于破译古埃及文字),因为该平台试图破译蛋白质的氨基酸序列的结构“含义”。Rosetta 出现7年之后,Rosetta@home 项目于2005年10月6日发布。许多参与到 Rosetta 起步工作的研究生和研究人员已经搬迁到各个不同的大学和研究机构。这使 Rosetta 项目的不同分支也得到加强。
RosettaDesign
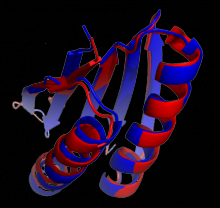
RosettaDesign 是基于 Rosetta 的一个蛋白质设计工具。它始于2000年对蛋白质G的折叠路径的一项研究。2002年,RosettaDesign 被用来设计 TOP7。TOP7 是一种93个氨基酸长度的 α/β 型蛋白质,具有在自然界中尚未被发现的整体折叠结构。这一全新的结构由 Rosetta 预测,与X射线晶体学确定的结构的之间的 RMSD 在1.2埃范围内,是一个精度极高的结构预测。Rosetta 和 RosettaDesign 最早设计和精确预测出这一长度的新型蛋白质,受到广泛的认可。他们2003年在《科学》杂志上发表的相关论文已被270多篇论文引用。这项研究的显著成果 TOP7 被选为蛋白质数据库2005年10月的“月度分子”(Molecule of the Month)。这项预测与其X射线晶体学结构的叠合被包含在 Rosetta@home 的 logo 设计中。曾在贝克实验室当博士后、现任北卡罗来纳大学教堂山分校助理教授的布赖恩·库尔曼提供了 RosettaDesign 的在线服务。
RosettaDock
RosettaDock 在2002年 CAPRI 实验时作为贝克实验室的蛋白质-蛋白质对接预测算法被添加到 Rosetta 软件套装中。在那次实验中,RosettaDock 对化脓性链球菌毒素A和T细胞受体β链的对接做出了高精度预测,对一种猪的α淀粉酶与相应骆驼抗体的复合体做出了中等精度预测。尽管 RosettaDock 方法只在七种可能中给出两种可接受精度的预测,这已经足以让它在那届 CAPRI 实验中名列19个预测方法中的第7位。
RosettaDock 的基础工作主要是由杰弗里·格雷在华盛顿大学期间完成的。后来他搬到约翰·霍普金斯大学接受另一个工作职位。因此,RosettaDock 在这之后的开发出现了两个分支。这两个分支在侧链建模、诱捕选择等方面存在细微差异。尽管存在这些差异,贝克和格雷的方法均在第二次 CAPRI 实验中表现出色,在30个小组中分列第5位和第7位。
2006年10月,RosettaDock 被集成到 Rosetta@home 中。这一方法首先仅用蛋白质骨架进行快速、粗略的对接建模,然后进行缓慢的全原子优化。在后一阶段中,两个互相作用的蛋白质之间的相对位置以及蛋白质-蛋白质界面的侧链交互作用同时被优化,从而得到最低能量构造。Rosetta@home 网络提供的巨大的运算能力,以及骨架灵活性和连接环建模经修订的折叠树表述,使 RosettaDock 在第三次CAPRI实验中名列63个预测组中的第6位。
Robetta
Robetta 是一个自动化的蛋白质结构预测服务,由贝克实验室提供,用于非商业性质的 ab initio 和比较建模。它自2002年起参加 CASP 实验,在当届 CASP5 的自动化服务器预测类别中名列前茅。此后,Robetta 又参加了 CASP6 和 CASP7,成绩比自动化服务器和人工预测组的平均水平都高。
到 CASP6 时为止,Robetta 构建蛋白质结构模型时采用的方法是,首先用 BLAST、PSI-BLAST 和 3D-Jury 搜索结构同源体,然后通过序列与 Pfam 数据库中的结构族的匹配,将目标序列解析为单独的结构域或者独立的折叠单元。下一步,具有结构同源体的结构域则要遵循一个基于模板的模型(即同源建模)协议。在此处,贝克实验室内部的一个程序 K*sync 会生成一组序列同源体,其中的每一项由 Rosetta 的 de novo 方法建模,产生诱捕(可能的结构)。然后,由低分辨率 Rosetta 能量函数确定的最低能量模型被选为最终的结构预测方案。对于未检测到结构同源体的结构域,将根据 de novo 协议,选定生成的诱捕中具有最低能量的模型作为最终的结构预测方案。这些结构域预测方案将被连接在一起,用来研究蛋白质内跨结构域、三级结构级别的交互作用。最后,根据一个蒙特·卡罗构造搜索协议来构建侧链贡献。
在 CASP8 中,由于 Rosetta 高分辨率全原子优化方法的引入,Robetta 的性能得到提高。而缺少这一方法被认为是 Robetta 在 CASP7 中精度低于 Rosetta@home 的主要原因。
Foldit
2008年5月9日,贝克实验室接受 Rosetta@home 用户关于交互式版本的建议,发布了 Foldit。这是一个基于 Rosetta 平台的在线蛋白质结构预测游戏。截至2009年1月9日,Foldit 的注册用户已经接近7.9万名。这个游戏赋予用户一系列的控制功能(如“摇动”、“摆动”、“重建”等),来操纵目标蛋白质的骨架和氨基酸侧链,以获得最佳能量构造。用户能够以单独或者集体的形式来进行游戏,通过改进结构预测方案来获得积分。用户还可以通过“决斗”功能来与其他用户进行竞赛,在20个动作内得到最低能量结构的用户获胜。